Door fysiologische omstandigheden na te bootsen, kunnen onderzoekers metalen bindmiddelen vinden
Onderzoekers hebben een methode ontwikkeld voor het identificeren van kleine moleculen die metaalionen binden. Metaalionen zijn essentieel in de biologie. Maar het kan een uitdaging zijn om te identificeren met welke moleculen – en vooral met welke kleine moleculen – deze metaalionen interageren.
Om metabolieten te scheiden voor analyse, maken conventionele metabolomics-methoden gebruik van organische oplosmiddelen en lage pH's, waardoor metaalcomplexen kunnen dissociëren. Pieter C. Dorrestein van de Universiteit van Californië in San Diego en collega's wilden de complexen bij elkaar houden voor analyse door de oorspronkelijke omstandigheden in cellen na te bootsen. Maar als ze tijdens de scheiding van moleculen fysiologische omstandigheden hadden gebruikt, hadden ze de scheidingsomstandigheden opnieuw moeten optimaliseren voor elke fysiologische toestand die ze wilden testen.
In plaats daarvan ontwikkelden de onderzoekers een aanpak in twee fasen die fysiologische omstandigheden introduceert tussen een conventionele chromatografische scheiding en een massaspectrometrische analyse (Nat. Chem. 2021, DOI: 10.1038/s41557-021-00803-1). Eerst scheidden ze een biologisch extract met behulp van conventionele hoogwaardige vloeistofchromatografie. Vervolgens pasten ze de pH aan van de stroom die de chromatografische kolom verliet om fysiologische omstandigheden na te bootsen, voegden metaalionen toe en analyseerden het mengsel met massaspectrometrie. Ze voerden de analyse twee keer uit om massaspectra van kleine moleculen met en zonder metalen te verkrijgen. Om te identificeren welke moleculen metalen binden, gebruikten ze een computationele methode die piekvormen gebruikt om verbindingen af te leiden tussen de spectra van gebonden en ongebonden versies.
Eén manier om fysiologische omstandigheden verder na te bootsen, zegt Dorrestein, zou zijn door hoge concentraties ionen zoals natrium of kalium en lage concentraties van het betreffende metaal toe te voegen. “Het wordt een concurrentie-experiment. Het zal je feitelijk vertellen: oké, dit molecuul heeft onder deze omstandigheden meer neiging om natrium en kalium te binden, of dit ene unieke metaal dat je hebt toegevoegd,' zegt Dorrestein. "We kunnen veel verschillende metalen tegelijkertijd infuseren, en we kunnen de voorkeur en selectiviteit in die context echt begrijpen."
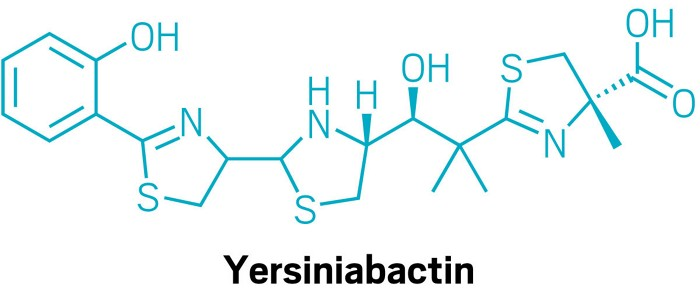
In kweekextracten van Escherichia coli identificeerden de onderzoekers bekende ijzerbindende verbindingen zoals yersiniabactine en aerobactine. In het geval van yersiniabactine ontdekten ze dat het ook zink kan binden.
De onderzoekers identificeerden metaalbindende verbindingen in monsters die zo complex zijn als opgelost organisch materiaal uit de oceaan. "Dat is absoluut een van de meest complexe monsters waar ik ooit naar heb gekeken", zegt Dorrestein. “Het is waarschijnlijk net zo complex als, zo niet complexer dan, ruwe olie.” De methode identificeerde domoïnezuur als een koperbindend molecuul en suggereerde dat het Cu2+ als een dimeer bindt.
“Een omics-aanpak om alle metaalbindende metabolieten in een monster te identificeren is uiterst nuttig vanwege het belang van biologische metaalchelatie”, schrijft Oliver Baars, die metaalbindende metabolieten bestudeert die door planten en microben worden geproduceerd aan de North Carolina State University. e-mail.
“Dorrestein en collega’s bieden een elegante, broodnodige test om beter te onderzoeken wat de fysiologische rol van metaalionen in de cel zou kunnen zijn”, schrijft Albert JR Heck, een pionier op het gebied van inheemse massaspectrometrieanalyses aan de Universiteit Utrecht, in een e-mail. “Een mogelijke volgende stap zou zijn om de metabolieten onder natuurlijke omstandigheden uit de cel te extraheren en deze ook onder natuurlijke omstandigheden te fractioneren, om te zien welke metabolieten welke endogene cellulaire metaalionen dragen.”
Chemisch en technisch nieuws
ISSN0009-2347
Auteursrecht © 2021 American Chemical Society
Posttijd: 23 december 2021

